抗がん剤スクリーニング支援のための基盤情報として、化合物の制がん作用と関連する遺伝子発現変化の情報を提供することを目的としている。有望な分子標的薬、抗がん剤などについて、マイクロアレイを用いた網羅的遺伝子発現解析により、化合物処理した際のがん細胞の発現プロファイルを取得し、制がん作用と関連する遺伝子発現変化をデータベース化する。
分子標的薬をはじめとする、典型的抗がん剤、典型的標準阻害剤について、約100種類の化合物を目標にデータ取得を進めている。これらについては、未処理コントロールとペアワイズに解析し、各薬剤処理によって発現変化する遺伝子の情報を順次公開していく。 これまでに取得されたデータを以下に公開する。
5種類のヒトがん細胞株(結腸がん HT-29,卵巣がん SKOV3,白血病 K562,肺がん H2228,肺がん PC-9)をさまざまな抗がん剤で6時間ないし16時間処理した。(薬剤処理濃度は、GI50値の3~10倍で、48時間接触で80%以上増殖阻害する濃度を基本とする。)その後、GeneChip Human Genome U133 Plus 2.0 Array (Affymetrix)を用い、マイクロアレイによる網羅的遺伝子発現解析を行い、変動遺伝子群(シグニチャー)を抽出した。
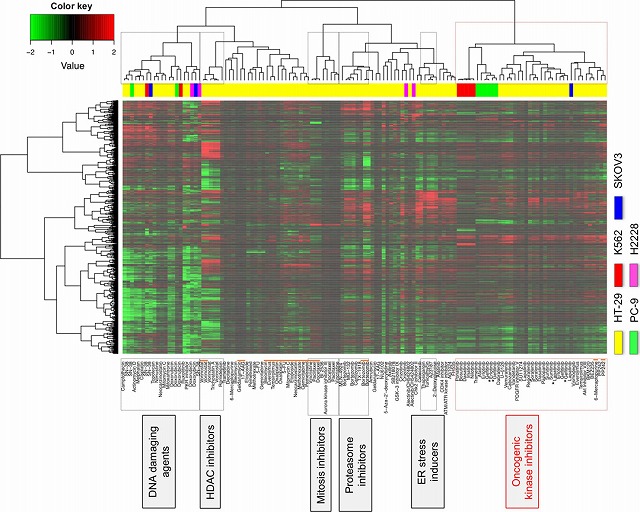
化合物のクラスター解析
取得された83の抗がん剤(129処理サンプル)の遺伝子発現データをもとに、クラスター解析を行った(Mashima et al. Cancer Science, 2015)。その結果、同じ作用機序をもつ化合物が概ね同じクラスターに分類された。
次に,HT29細胞株に対して38のキナーゼ阻害剤を作用させたときの遺伝子発現変動に対して同様にクラスター解析を行ったところ,標的経路ごとに分類された.
そこでさらに、作用機序ごとに特異的に高発現または低発現する遺伝子群の探索を行った。下表にその遺伝子リストを示す。
| Mode of action | up-regulated | down-regulated |
|---|---|---|
| Classical DNA damaging agents (16化合物) |
DNAdamage_up.csv | DNAdamage_down.csv |
| HDAC inhibitors (5) | HDACi_up.csv | HDACi_down.csv |
| Tubulin binding agents (6) | Tubulin_up.csv | Tubulin_down.csv |
| proteasome inhibitors (5) | proteasomei_up.csv | proteasomei_down.csv |
| ER stress inducers (4) | ERstress_up.csv | ERstress_down.csv |
| Raf/MEK/ERK inhibitors (6) | RAF_up.csv | RAF_down.csv |
| PI3K/AKT/mTOR inhibitors (7) | PI3K_down.csv |
今回構築したデータベースとConnectivity Mapで用いられているデータ処理法(Lamb et.al. Science 2006)を用いて、遺伝子発現パターンから化合物の作用機序を予測するシステムを構築した(Ushijima et.al. Cancer Science 2013)。これを用いることで、自分の手元にある作用機序が未知の抗がん剤候補化合物について、既存の抗がん剤との類似性を検討することができる。
オンライン版はこちら(genomecenter.jfcr.or.jpにジャンプします)
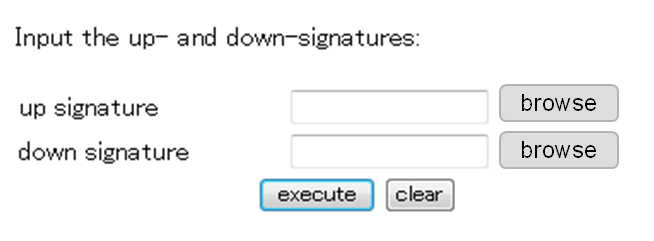
ここでは、Affymetrix GeneChipR Human Genome U133 Plus 2.0 Arrayを使用して実験データを取得した場合の、解析用ファイル作成の手順を示す。
(1) 遺伝子発現を測定した任意のデータ(例:化合物Xを処理した培養がん細胞と未処理の細胞)を比較し、有意に発現量の増加したprobe set(up tag)と減少したprobe set(down tag)を同定する。
(2) Microsoft Excelなどを使用し、同定したsignatureのprobe set ID(例:200800_s_at)のみを1行に1つずつ1列に並べたリストを、upとdownとで別々に作成する.?
(3) 作成したリストをタブ区切り形式のテキストファイルとして保存する. ファイルはup tag fileとdown tag fileの2つを作成する.
(4) Connectivity scoring analysis の解析サイトに入り、画面上の"up signature" と"down signature"ボタンをクリックし、それぞれのファイルをアップロードする.
(5) 画面上の"execute" ボタンをクリックする。(結果は、入力された化合物Xが変動させる遺伝子群とより類似性の高い遺伝子群を誘導する抗がん剤の順にリストとして表示される。)
入力ファイルの例: GR033-up3-300.txt (up) GR033-down3-300.txt (down)
*今回構築したConnectivity scoring analysisやKEGG pathway解析を行うためのプログラムのダウンロードも可能である(専門家用プログラムを参照)。